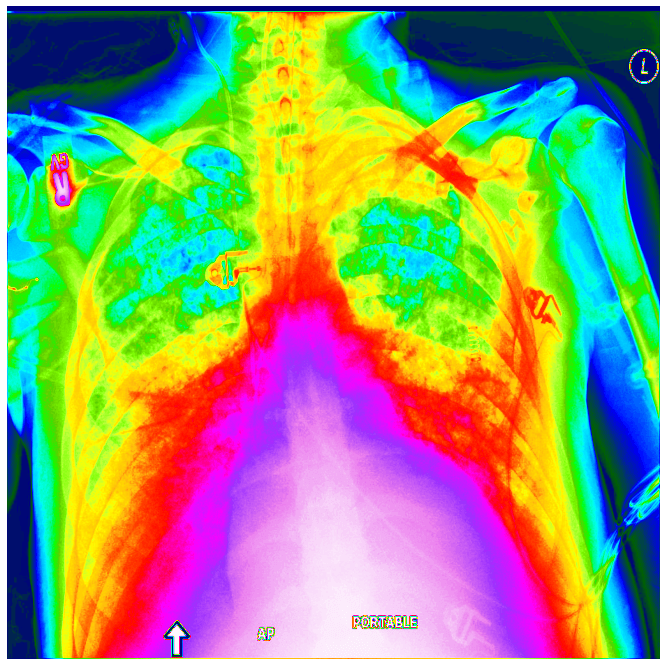
ในการพัฒนาระบบ AI สำหรับวินิจฉัยโรค ชนิดของข้อมูล Dataset ที่เราจะพบบ่อย ๆ หนึ่งในนั้นคือไฟล์ DICOM ซึ่งเป็นไฟล์ภาพถ่ายทางการแพทย์ Mdical Imaging สำหรับฟิล์ม X-Ray, MRI, CT-Scan, Ultrasound ใน ep นี้เราจะมาทำความเข้าใจ ไฟล์ DICOM กันให้มากขึ้น
DICOM คืออะไร

DICOM ย่อมาจาก Digital Imaging and Communications in Medicine เป็นมาตรฐานกลางในการสื่อสาร จัดเก็บ เรียกดู ประมวลผล จัดพิมพ์ และแสดงผลข้อมูลภาพถ่ายทางการแพทย์ Mdical Imaging กำหนดโดย องค์กรชื่อ National Electrical Manufacturers Association (NEMA)
มาตรฐาน DICOM ทำให้เราสามารถใช้ข้อมูลภาพถ่ายทางการแพทย์ Mdical Imaging ต่าง ๆ ร่วมกันได้ข้ามระบบ ระหว่างอุปกรณ์ต่าง ๆ ตั้งแต่ อุปกรณ์ถ่ายภาพ, PACS (Picture Archiving and Communication System), Workstation, VNAs และเครื่องพิมพ์ จากหลากหลายยี่ห้อ
ภายในไฟล์จะประกอบด้วยทั้งส่วน Header ที่เป็น Metada และส่วนของข้อมูลรูปภาพ ซึ่งมีได้มากกว่า 1 รูปต่อ 1 ไฟล์ เรามาดูตัวอย่างไฟล์ และรูปแบบข้อมูลในไฟล์กันดีกว่า
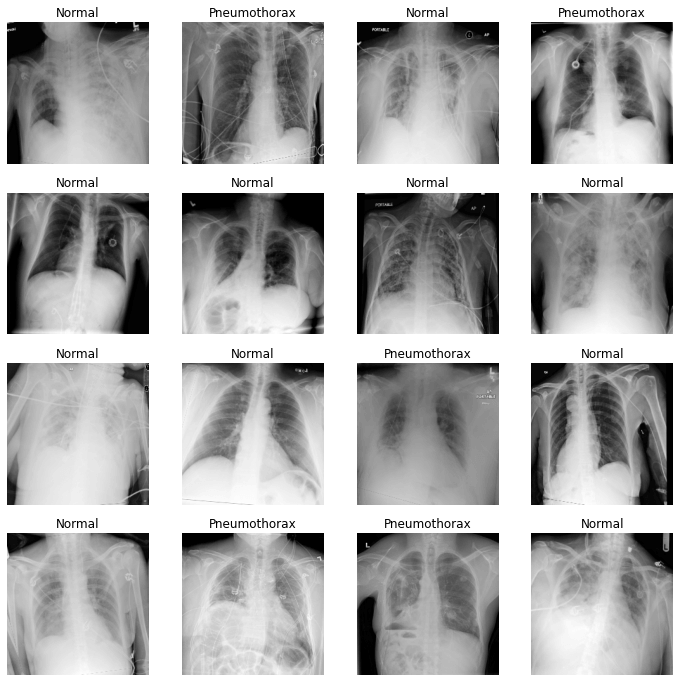
Weighted Cross Entropy Loss
ในเคสนี้จำนวนข้อมูลตัวอย่าง ในแต่ละ Class แตกต่างกันมาก เรียกว่า Class Imbalance เราจะใช้ Loss Function แบบใหม่ ชื่อว่า Weighted Cross Entropy Loss