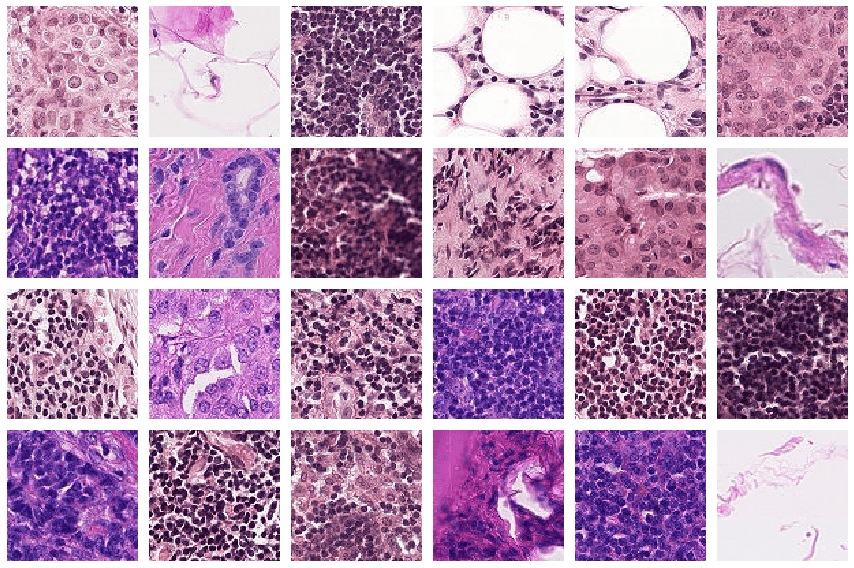
ใน ep นี้ เราจะมาสร้างโมเดลที่ใช้จำแนก โรคมะเร็งระยะลุกลาม Metastatic Cancer จากรูปภาพ Patch เล็ก ๆ ของ Whole Slide Imaging ที่ตัดมาจาก Digital Pathology Scans รูปใหญ่
มะเร็งระยะลุกลาม (Metastatic Cancer) คืออะไร
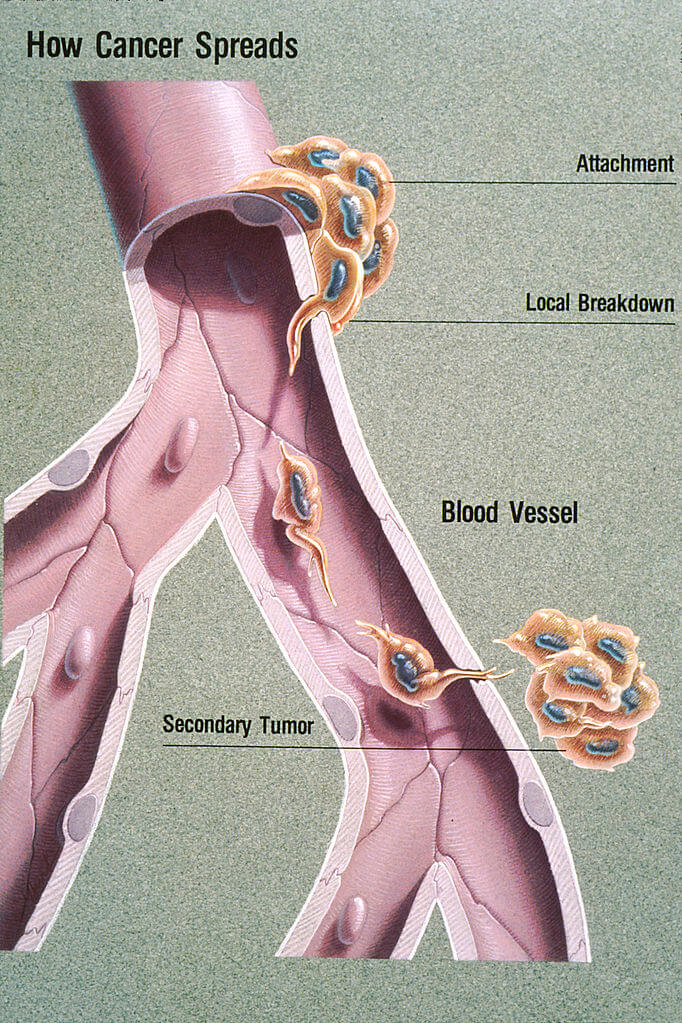
Metastatic Cancer หรือ มะเร็งระยะลุกลาม คือ การที่เซลล์มะเร็งแพร่กระจายลุกลามไปยังอวัยวะอื่น ๆ ภายในร่างกาย โดยมักจะแพร่กระจายไปทางต่อมน้ำเหลือง และกระแสเลือด Metastatic Cancer หรือ Metastatic Tumor คือรูปแบบหนึ่งของการแพร่กระจายจากเซลล์มะเร็งต้นตอ ไปยังส่วนต่าง ๆ ของร่างกาย
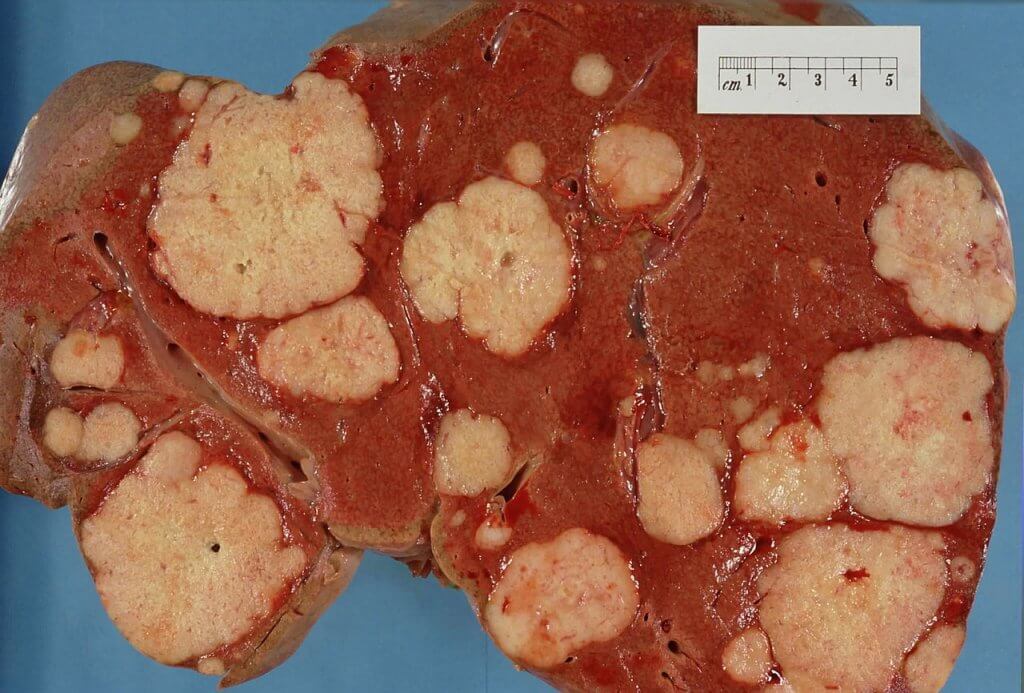
Metastasis คืออะไร
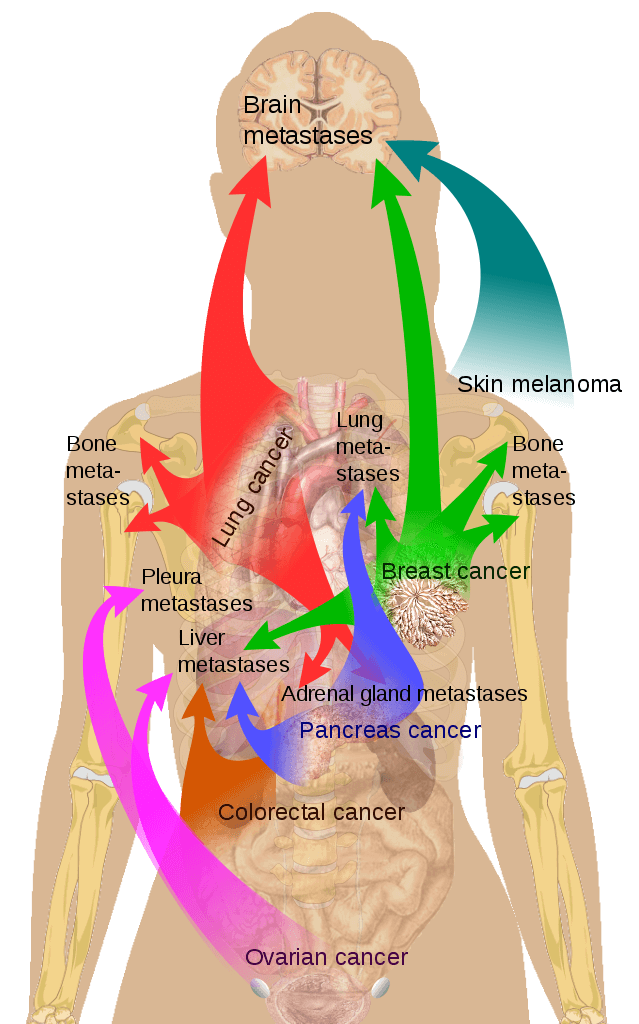
Metastasis คือ ตัวแพร่จากเซลล์มะเร็งต้นตอ ไปยังส่วนอื่น ๆ ของร่างการผู้ป่วย ส่วนของอวัยวะใหม่ที่ติดโรคมะเร็งจะเรียกว่า Metastases (Mets) ที่มักจะถูกจำแนก แยกจากเนื้องอกมะเร็ง Cancer Invasion ที่ขยายขึ้นโดยตรง และลุกลามไปยังเซลล์เนื้อเยื่อรอบ ๆ บริเวณเดียวกัน
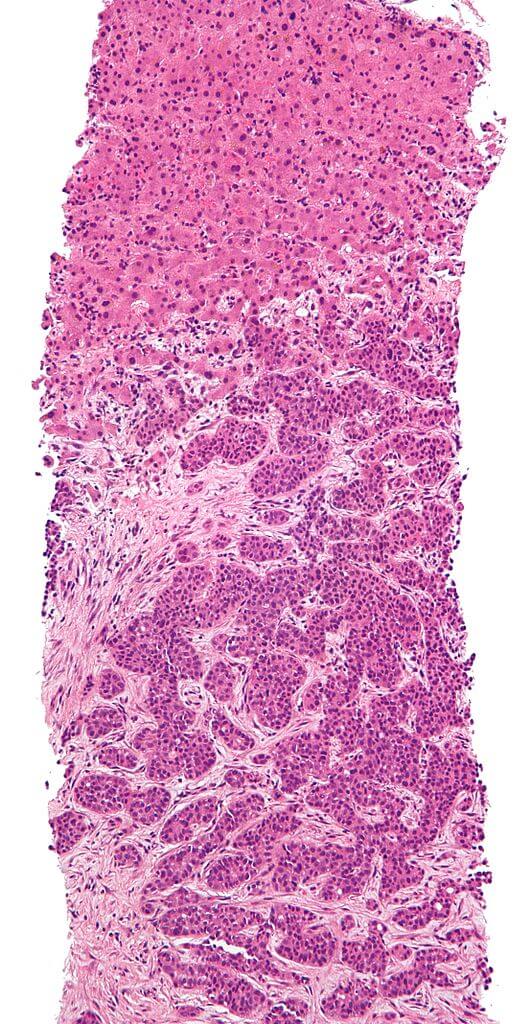
ข้อมูล Whole Slide Imaging (WSI) ใน Dataset จาก Kaggle นี้มาจาก PatchCamelyon (PCam) benchmark dataset
Ranger Optimizer
จากที่ ep ก่อน ๆ ใช้ Optimizer ชื่อ Adam ใน ep นี้ เราจะเปลี่ยนไปใช้ Ranger ที่มาจากการรวม RAdam และ Look Ahead จะอธิบายต่อไป
DenseNet Architecture
จากที่ ep ก่อน ๆ ใช้ Model Architecture ชื่อ ResNet ใน ep นี้ เราจะเปลี่ยนไปใช้ DenseNet ที่ซับซ้อนมากขึ้น จะอธิบายต่อไป
Cross Entropy Loss + Label Smoothing
Loss Function เราจะใช้ Label Smoothing Cross Entropy Loss Function
Test-Time Augmentation (TTA)
ในการวินิจฉัย เราจะใช้เทคนิค Test-Time Augmentation (TTA) เป็นการนำเทคนิค Data Augmentation มาใช้กับ Test Set มาช่วยเพิ่มความแม่นยำ จะอธิบายต่อไป