โรคมะเร็งผิวหนัง นับเป็นปัญหาใหญ่ในทางสาธารณสุข ทุก ๆ ปี ในประเทศสหรัฐอเมริกา เราจะพบผู้ป่วยใหม่ มากกว่า 5 ล้านราย
มะเร็งผิวหนัง Melanoma เป็นมะเร็งผิวหนังชนิดที่ร้ายแรงที่สุด เป็นมะเร็งผิวหนังชนิดที่คร่าชีวิตคนมากที่สุด ในปี 2015 ทั่วโลก มีการตรวจพบ Melanoma มากกว่า 350,000 เคส โดยมีผู้ป่วยเสียชีวิต 60,000 คน ถึงแม้อัตราการเสียชีวิตจะสูง แต่ถ้ามีการวินิจฉัยโรคมะเร็งผิวหนังที่ง่ายขึ้น ตรวจพบตั้งแต่ระยะเริ่มต้น และรักษาได้อย่างทันท่วงที เราจะสามารถเพิ่มอัตราการรอดชีวิต ได้มากกว่า 95%
ใน ep นี้ เราจะมาสร้าง AI โมเดล Deep Learning ที่จะวินัจฉัยโรคมะเร็งผิวหนัง ด้วยการจำแนกรูปถ่ายผิวพรรณ ที่มีความผิดปกติของเม็ดสี ว่าเป็นโรคอะไรใน 7 โรคที่กำหนด ด้วยความแม่นยำ 94%
HAM10000 Dataset
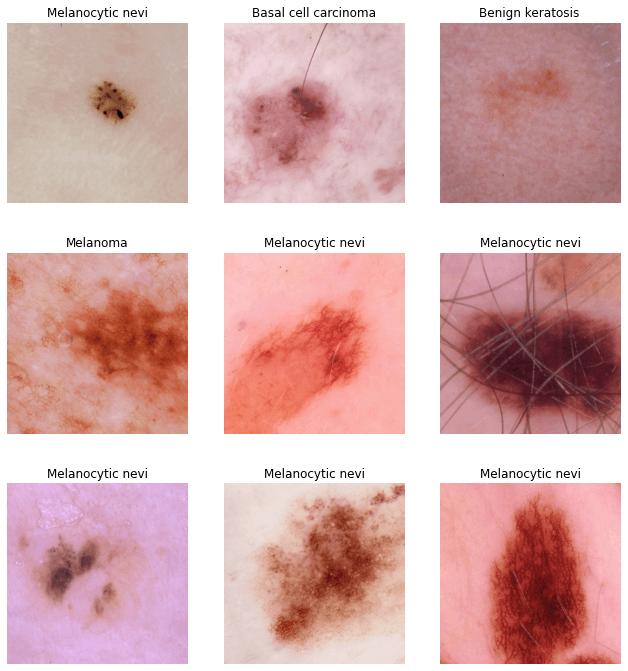
HAM10000 (“Human Against Machine with 10000 training images”) ชุดข้อมูลรูปผิวหนัง จากประชากรกลุ่มต่าง ๆ ประกอบด้วย 10,015 รูป สำหรับเป็น Training Set ในการสร้างโมเดล Machine Learning เพื่อการศึกษาและวิจัย
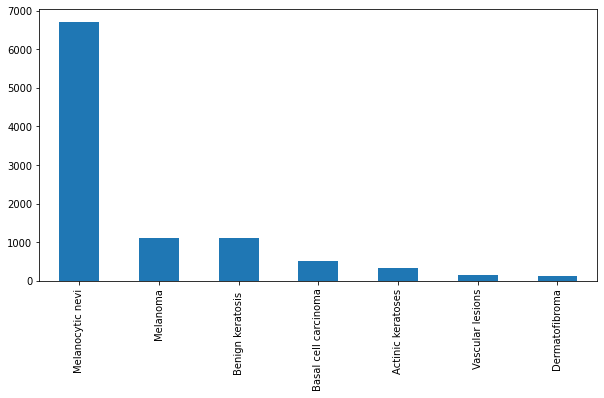
ในชุดข้อมูลประกอบด้วย ตัวอย่างเคสแผลผิวหนังที่เม็ดสีผิดปกติ แบบต่าง ๆ ได้แก่ Actinic keratoses and intraepithelial carcinoma / Bowen’s disease (akiec), basal cell carcinoma (bcc), benign keratosis-like lesions (solar lentigines / seborrheic keratoses and lichen-planus like keratoses, bkl), dermatofibroma (df), melanoma (mel), melanocytic nevi (nv) and vascular lesions (angiomas, angiokeratomas, pyogenic granulomas and hemorrhage, vasc).
รายละเอียดเพิ่มเติม ใน ep ที่แล้ว AI จำแนกปัญหาผิวพรรณ
Focal Loss คืออะไร
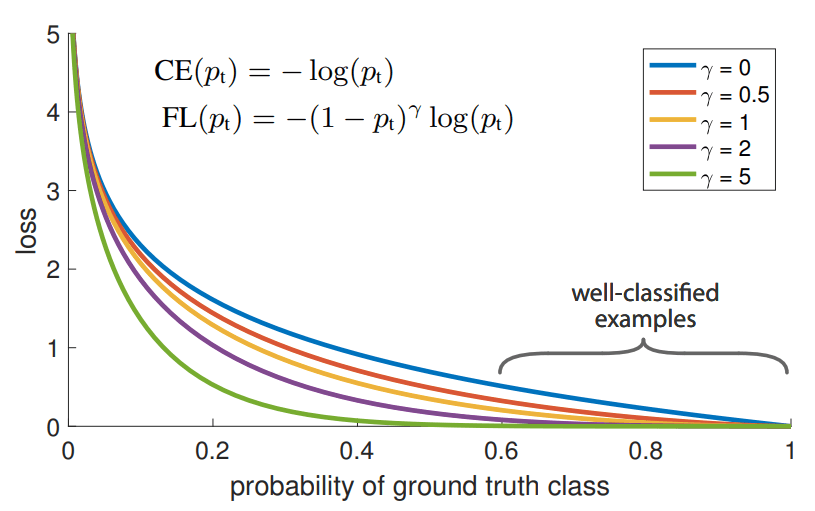
เนื่องจากจำนวนข้อมูลตัวอย่าง แต่ละ Class แตกต่างกันมาก เรียกว่า Class Imbalance แทนที่เราจะใช้ Cross Entropy Loss ตามปกติที่เรามักจะใช้ในงาน Classification ในเคสนี้เราจะเปลี่ยนไปใช้ Loss Function พิเศษ ที่ออกแบบมาเพื่อแก้ปัญหานี้ เรียกว่า Focal Loss ดังสมการด้านล่าง
\(\text{FL}(p_t) = -\alpha_t (1 – p_t)^{\gamma} \, \text{log}(p_t)\)รายละเอียดเพิ่มเติม Focal Loss คืออะไร